La evaluación histológica de las secciones intestinales mencionadas en el artículo previo debería permitir la sospecha del posible agente involucrado en el proceso y, en algunos casos, definir la presencia de agentes infecciosos. Por ejemplo, la atrofia de las vellosidades puede estar causada por coccidios (I. suis), Rotavirus, GETv, PEDv, Deltacoronavirus o incluso C. perfringens tipo A. Si las muestras pertenecen a lechones de más de cinco o seis días, podemos encontrar formas sexuales o asexuales de I. suis en el citoplasma de los enterocitos del extremo de las vellosidades. Si no se encuentran coccidios, puede realizarse inmunohistoquímica para detectar la presencia de antígenos víricos de Rotavirus o Coronavirus en los enterocitos. En casi todos los casos de ETEC, el examen histológico permitirá la detección de una miríada de organismos cocobacilares adheridos a la superficie del enterocito en la base o en la pared lateral de las vellosidades. C. perfringens tipo C causará una lesión transmural fibrinonecrótica y hemorrágica de distribución segmentaria en el intestino delgado. C. difficile es el único agente infeccioso que causará lesiones en el intestino grueso. El edema de mesocolon se observa a menudo. Los principales hallazgos en casos de infección por C. difficile son la reducción de las células caliciformes y una gran infiltración de neutrófilos en la mucosa del colon que, en algunos casos, rompen el epitelio como una erupción volcánica. C. perfringens tipo A es el más difícil de determinar por histopatología, ya que puede causar atrofia de las vellosidades, pero el hecho de no encontrar esta lesión no descarta dicha posibilidad.
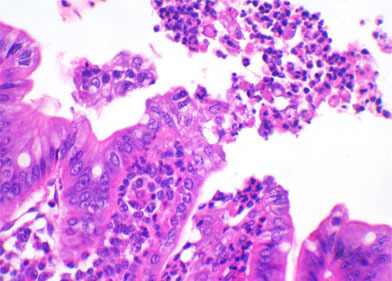
Clostridium difficile
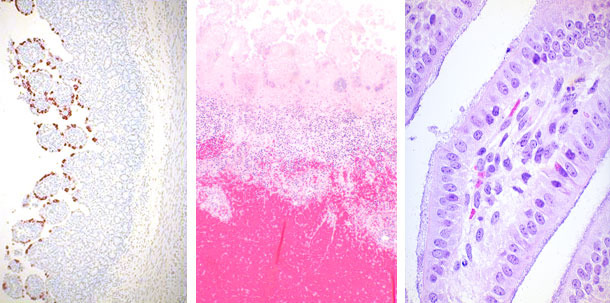
| Rotavirus | Clostridium perfringens | E. coli |
Es importante que la bacteriología de muestras frescas de intestino se lleve a cabo tanto en condiciones aeróbicas como anaeróbicas. E. coli crecerá fácilmente en agar sangre y MacConkey pero incluso el crecimiento puro de colonias beta-hemolíticas no es un diagnóstico definitivo ya que se requiere tipificación para demostrar la presencia de factores patogénicos, como genes de fimbrias (F4, F5, F41, F18,…) y toxinas (LT, STa, STb, STx2e, …). Esta tipificación a menudo se lleva a cabo mediante un multiplex PCR. Para el diagnóstico de C. perfringens tipo C es esencial el cultivo anaeróbico, junto a la tipificación molecular (multiplex PCR) para la detección de genes de toxinas, como beta toxina. Las rutinas bacteriológicas para C. perfringens tipo A y C son idénticas. En el pasado, la detección del gen para la toxina beta-2 era el marcador para las cepas patogénicas de C. perfringens tipo A, pero estudios recientes de nuestro grupo y del grupo de la Iowa State University han demostrado que las cepas beta-2 positivas de C. perfringens tipo A son más frecuentes en animales sanos que en cerdos diarreicos. Como resultado, la definición final y el diagnóstico de C. perfringens tipo A es el más complicado.
El aislamiento viral es difícil en el diagnóstico rutinario, por lo que se hacen tests alternativos. Casi nunca se realiza microscopía electrónica de transmisión para la detección de corona o rotavirus y sólo se usa en algunos laboratorios diagnósticos de EE.UU. Con mayor frecuencia se realiza electroforesis en gel de poliacrilamida (PAGE) en muestras fecales para la identificación del RNA segmentado de rotavirus A, B y C, aunque su sensibilidad es mucho mayor si se utilizan PCRs multiplex. Para el diagnóstico de coronavirus (PED, GET y Deltacoronavirus), en Norte América, se ha utilizado muy a menudo la técnica PCR multiplex, utilizando un conjunto distinto de cebadores.
El material fecal del intestino grueso es valioso para la detección de las toxinas A y B de C. difficile. Como que este agente forma parte de la microbiota intestinal normal, para su diagnóstico es esencial la detección de las mencionadas toxinas mediante los kits comerciales de ELISA. Es importante mencionar que estos kits comerciales de ELISA han sido desarrollados y optimizados para muestras humanas y que la sensibilidad en heces porcinas es baja. Por lo tanto, el tamaño de la muestra es importante y se recomienda analizar por lo menos 3-6 muestras fecales de animales diarreicos. En base al mismo principio, el examen directo de heces se utiliza a menudo para la detección de oocitos de coccidios; sin embargo, como la sensibilidad también es baja, la recomendación es recoger muestras de 3-5 lechones por camada con diarrea clínica, en el 10 % de las camadas entre 7 y 21 días.
Puede concluirse que la detección de agentes enteropatógenos en lechones recién nacidos siempre es complicada. El principal punto que no debe olvidarse es el enfoque global necesario para evaluar todas las posibilidades. En este sentido, deben tomarse muestras y analizarse frente a todos los agentes que puedan causar dicha diarrea. La PCR es una herramienta potente, aunque no hay que apostarlo todo a esta carta. Técnicas más antiguas, como la histopatología pueden guiar la interpretación de los resultados y ampliar tus perspectivas.






